最近看了几篇关于共线性分析的微信/简书推送,发现不少研究人员把“编码基因共线性”错误地描述为“全基因组共线性”。这是两个完全不同的概念,前者主要是基于蛋白水平,蛋白的保守性很高,即使分化较远的物种间也能找到同源基因片段;而后者则是基于DNA水平,其中包含了大量的非编码片段,变异程度即使是在近缘物种间也非常大。那么,接下来就好好聊一聊什么是共线性?为什么进行全基因组共线性分析?以及如何进行全基因组共线性分析?
1. 共线性
所谓的共线性主要是用来描述同一染色体上基因的位置关系,也就是指由同一祖先型分化而来的不同物种间基因的类型以及相对顺序的保守性(即基因的同源性+基因的排列顺序)。共线性片段的大小与物种之间的分化时间有很大关系:分化时间较短的物种间,积累的变异较少,会保留更多从祖先遗传下来的特征;相反的,分化时间较长的物种间由于变异积累而导致共有的特征变少,反而获得较短的共线性片段。此外,基因同源又可以分为直系同源和旁系同源。直系同源基因指存在于祖先基因组中,随后因为物种分化,分别遗传给不同的后代,这些基因在结构和功能上有很高的相似性。旁系同源基因指同一基因组中由于基因复制而产生的的同源基因,这些基因往往变异较大,从而可能出现功能变异。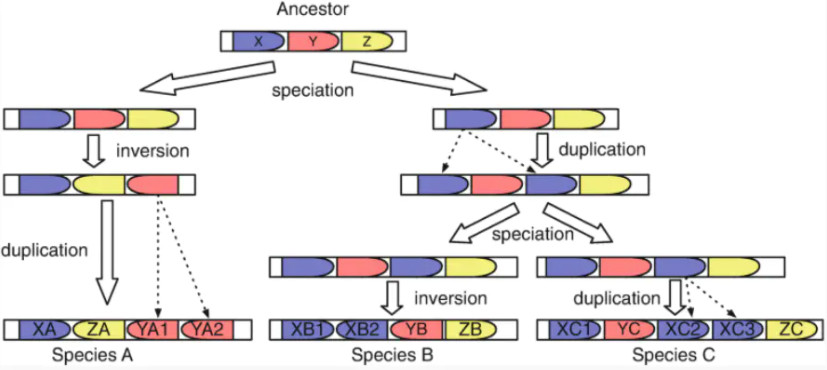
2.共线性分析的应用
测序发展初期,人们只能测得部分序列,但这些数据量少,不利于全面分析基因功能。另外,单个物种基因组序列也无法完整的描述该物种的全面信息,而且也无法挖掘真正的进化事件,比如基因丢失、基因获得等。近几年,随着测序技术的快速发展,为大规模的全基因组测序创造了很好的条件。而比较基因组的出现,更是进一步推动了近缘物种或个体的全基因组测序,其中比较基因组中很大的一部分工作就是集中在全基因组比对上。
共线性分析是比较基因组中必不可少的分析策略,因为它允许分析物种间大尺度和小尺度的分子进化事件。大尺度进化事件主要包括对基因组内重排和复制事件的估计,例如,可以使用人与小鼠之间的全基因组比对来识别共线性同源区块,然后确定重排事件,从而解释两个基因组结构差异。小尺度进化事件则是针对基因组水平的碱基替换速率以及插入、缺失事件。从共线性片段中可以识别出的物种间小尺度和大尺度突变事件,这些都可以作为物种树推断数据。此外,结合构建的两个尺度的基因组进化模型,共线性比对还能够完成祖先基因组重建的任务。
由于基因组共线性通常可以预测同源序列,并且同源序列可能具有相似的功能,因此全基因组共线性分析对于功能预测是十分有价值的。可以通过在整个基因组的核苷酸水平上对齐,从而可以帮助预测编码和非编码区域的功能。 例如,如果我们对人类基因组中特定的疾病相关区域感兴趣,我们可能会使用对齐来识别其小鼠同源基因的位置。 通过对小鼠同源片段的了解将使我们能够更好地了解这个基因组区域的进化历史,并可能进行遗传操作实验。
3.基因组共线性比对工具
全基因组比对需要消耗的计算机内存很大,而且运行时间较长。另外,由于基因组复制事件广泛存在,尤其是植物基因组,可能无法很好地区分旁系同源基因,容易产生大量的假阳性比对结果。因此,选择合适的基因组比对软件尤其重要。目前比较成熟的基于全基因组共线性比对分析的软件有:MUMmer、progressiveMauve、Mugsy、LAST、Lastz、Cactus。
MUMmer MUMmer是一种非常快速的pairwise基因组比对工具,能够在四个小时内完成人类和黑猩猩的全基因组比对。它是通过使用后缀树数据结构来找到两个基因组之间的所有最大唯一匹配,从而达到了这一速度。MUMmer适用于非常近缘物种之间的基因组比对,尽管速度较快,但其灵敏度比LAST/lastZ要低。
**progressiveMauve progressiveMauve是一款以java编写的全基因组比对软件,具有可视化界面,便于之间查看比对结果,比对效果较好适用于细菌基因组的比对。此外,需要注意的是progressiveMauve采用的比对策略是无参比对。
Mugsy Mugsy比对速度快,效率高,而且比对的长度较长,但主要适用于近缘物种之间的比较,对于进化距离较远的物种比对效果不太理想。
LAST LAST的优势在于能够处理较大基因组(如脊椎动物基因组)之间的比较,而且具有很快的比对速度。但是,它目前还无法进行有gap的比对,这也在一定程度上导致它的比对长度较短,数量较多。另外,LAST能够有效的处理含有重复序列的区域,因为它采用的是可变长度的种子序列,大大改善了比对的效率。
Lastz Lastz相比于LAST而言,它能够允许基因组中少量gap的存在,比对长度较长,适用远缘物种之间的全基因组比对(如脊椎动物),其灵敏度稍稍低于LAST,速度也要比LAST慢一些。
Cactus Cactus主程序其实是打包了Lastz,但是它自身又采用了无参比对的策略,可以直接重构祖先的染色体序列。
值得推荐的软件: LAST、Lastz、Cactus
参考链接:https://link.springer.com/protocol/10.1007/978-1-4939-9074-0_4